Omics
La tecnología genómica permite un muestreo rápido y rentable para obtener mejores resultados
DESPLÁCESE PARA SABER MÁS
Quiénes somos
Los científicos del AOML utilizan el término ómico para referirse al estudio de varios campos como la genómica, la metagenómica, la metatranscriptómica, la proteómica, la metabolómica, la epigenómica y la secuenciación de amplicones de alto rendimiento. Estos campos emergentes nos ayudan a responder a preguntas de investigación sobre el ADN, el ARN, las proteínas y otras pequeñas moléculas de los organismos y el medio ambiente. Con esta información, podemos identificar los mecanismos que mantienen los sistemas marinos sanos y productivos.
Los cinco pilares de las actividades ómicas de la AOML
El programa Omics en el AOML trabaja para promover la resiliencia de los corales, desarrollar y transferir tecnologías emergentes, avanzar en las aplicaciones Omics para la pesca y el microbioma, y fomentar las capacidades bioinformáticas y de infraestructura de las que dependen todas las investigaciones y operaciones Omics. Este trabajo involucra a expertos en corales, pesquerías y microbiología en toda la agencia y a través del compromiso internacional.
Leer más noticias
Restauración de corales Ómicos
Resistencia y resiliencia
Nuestra investigación pretende identificar qué hace que ciertos corales sean más resistentes a factores de estrés como el calor, la acidificación de los océanos y las enfermedades. La comprensión de los fundamentos moleculares de la resistencia y la susceptibilidad de los corales, mediante la identificación de genotipos resistentes, permite a los gestores de recursos ser más eficaces en sus planes de restauración, especialmente a la hora de plantar corales en el arrecife.
Resistencia al estrés térmico
El AOML está trabajando en la identificación de genotipos de coral resistentes para aislar los mecanismos moleculares por los que la resistencia se transfiere a través de las colonias. La investigación en profundidad sobre el terreno de los patrones de blanqueo de los corales ha revelado que algunos corales son más resistentes al blanqueo por estrés térmico.
Esos corales tienen firmas genéticas particulares que se han aclimatado a +1,0°C más calientes que otros de la misma especie. Los investigadores podrían utilizar esta información para ayudar a construir arrecifes más resistentes para el futuro.
La identificación de marcadores genéticos de la resistencia de los corales, tanto mediante investigaciones de laboratorio como de campo, nos permitirá comprender las respuestas al blanqueo y ser proactivos cuando los factores de estrés ambiental afecten a nuestros ecosistemas arrecifales.
Resistencia a las enfermedades
Parte de nuestra investigación aborda las devastadoras consecuencias de la propagación de enfermedades por el sur de Florida y los Cayos de Florida a nivel molecular. Los científicos están modelando cómo se propaga la enfermedad a través de un arrecife para mejorar su predicción de la propagación de la enfermedad.
Con esta información, los investigadores pueden ayudar a mejorar la resistencia del arrecife y evaluar la longevidad de los distintos tipos de corales en él. El rastreo de la fuente microbiana también se está utilizando para rastrear las fuentes terrestres de contaminación, y la secuenciación del ADN se está utilizando para investigar la biodiversidad de los microbiomas de los corales.
Para entender el microbioma
Existe una necesidad global de indicadores de bajo coste que midan la salud de los ecosistemas marinos. El microbioma constituye la base de la red alimentaria y controla aspectos como el carbono, los nutrientes, los metales y las toxinas, lo que significa que no podemos comprender realmente el estado de los ecosistemas marinos hasta que no tengamos un buen conocimiento de su microbioma. Esta comprensión nos ha permitido apreciar el papel de los microbios en todos los aspectos de la vida, incluida la resistencia de los océanos y la gestión de los recursos marinos, así como las infecciones y las enfermedades.
Para detectar niveles tróficos superiores
Dado que el ADNe procede de células desprendidas o excretadas de un organismo marino, el ADNe ofrece una oportunidad única para detectar qué organismos marinos han estado en la zona utilizando únicamente muestras de agua de mar. Esto puede ser especialmente útil en zonas remotas y sensibles. También ofrece la posibilidad de detectar múltiples niveles tróficos a partir de una sola muestra.
Estamos trabajando en el uso del ADN ambiental (ADNe), es decir, el ADN del agua de mar filtrada, para entender qué tipos de animales de nivel trófico superior (como peces o mamíferos) se encuentran en una zona determinada. El ADNe se recoge del agua de mar o de los sedimentos, en lugar de los tejidos, de modo que las muestras pueden recogerse sin necesidad de capturar animales, procesar tejidos o arrastrarlos por hábitats sensibles.
Serie de vídeos sobre el ADN medioambiental
La investigación con ADN ambiental o "eDNA" es un área de la ciencia apasionante y emergente que puede ayudar a los científicos a gestionar las especies en peligro de extinción, las especies invasoras y a controlar la biodiversidad de los ecosistemas. Aprenda sobre el ADN ambiental a través de la serie de vídeos "Explorando el ADN ambiental" que cubre lo que es el "eDNA", la tecnología de muestreo de ADN ambiental desarrollada en el AOML de la NOAA, y una actividad práctica para la extracción de ADN.
eDNA para las tortugas marinas protegidas
Cumplimiento de los requisitos de vigilancia con eficiencia y mínimo impacto
Una población de ~60 tortugas verdes "negras"(Chelonia mydas agassizii) fue incluida en la lista de especies en peligro de extinción y ha sido monitoreada desde la década de 1970 en virtud de la Ley de Especies en Peligro de Extinción en el Refugio Nacional de Vida Silvestre del Sur de la Bahía de San Diego. Es necesario un seguimiento regular y continuado del tamaño de esta población para apoyar el plan de recuperación del Servicio Nacional de Pesquerías Marinas para las poblaciones de tortugas verdes del Pacífico de EE.UU. bajo un uso cambiante del hábitat. Los métodos tradicionales de seguimiento son de alto impacto, laboriosos y costosos. Utilizando la tecnología de ADN electrónico, puede ser posible un mapeo espacial de bajo impacto.
Echa un vistazo a la historia de la web publicada en la Investigación de la NOAA.

Genómica pesquera
Nuestra capacidad para gestionar eficazmente las pesquerías está limitada por nuestro conocimiento de las poblaciones pesqueras y su dependencia de las condiciones ambientales. La inclusión de la información genómica en la gestión pesquera puede mejorar el proceso de toma de decisiones y, por tanto, la sostenibilidad de las pesquerías.
La forma en que la información genómica afecta a las decisiones se evaluará utilizando técnicas avanzadas de evaluación de poblaciones y el marco de evaluación de estrategias de gestión adoptado por el Servicio Nacional de Pesquerías Marinas. El AOML y el Centro de Ciencias Pesqueras del Sureste (SEFSC) han formado una asociación de colaboración con un camino claro para la transición de los resultados a los planes de gestión de la pesca para los siguientes proyectos:
- Utilización de polimorfismos de un solo nucleótido (SNP de ADN) para diferenciar entre las poblaciones orientales y occidentales de atún rojo del Atlántico con el fin de informar sobre las evaluaciones de las poblaciones y la evaluación de las estrategias de gestión.
- Utilización de la secuenciación del ADN asociada a sitios de restricción (ddRAD-Seq) para acceder a la estructura de la población de la caballa real con el fin de abordar las incertidumbres apremiantes que afectan a la evaluación de las poblaciones de esta especie.
- Se están utilizando modelos empíricos para evaluar si los datos del microbioma (a veces denominado "eDNA") mejoran la capacidad de predecir las distribuciones de los peces y de comprender las interacciones tróficas y el uso del hábitat.
Mejora de la mano de obra y la infraestructura de la ómica
Laboratorio experimental de arrecifes
El Laboratorio Experimental de Arrecifes, situado en el campus de Virginia Key de la Universidad de Miami, en la Escuela Rosenstiel de Ciencias Marinas y Atmosféricas, se completó en septiembre de 2016. Esta instalación experimental única fue diseñada para estudiar el efecto combinado del estrés térmico y la acidificación de los océanos en los corales, de modo que los científicos puedan ver cómo responden los organismos coralinos a nivel molecular (ADN y ARN) en las condiciones actuales y en las posibles futuras.
Pase el ratón sobre los puntos para obtener más información o visite la página del Laboratorio Experimental de Arrecifes.
Con la construcción de instalaciones propias como éstas, podemos colmar lagunas críticas en materia de equipamiento, aumentar la eficacia y la integridad del procesamiento de muestras y ofrecer un lugar para que los socios (como el Southeast Fisheries Science Center) y otros colaboren en nuevos proyectos de investigación.
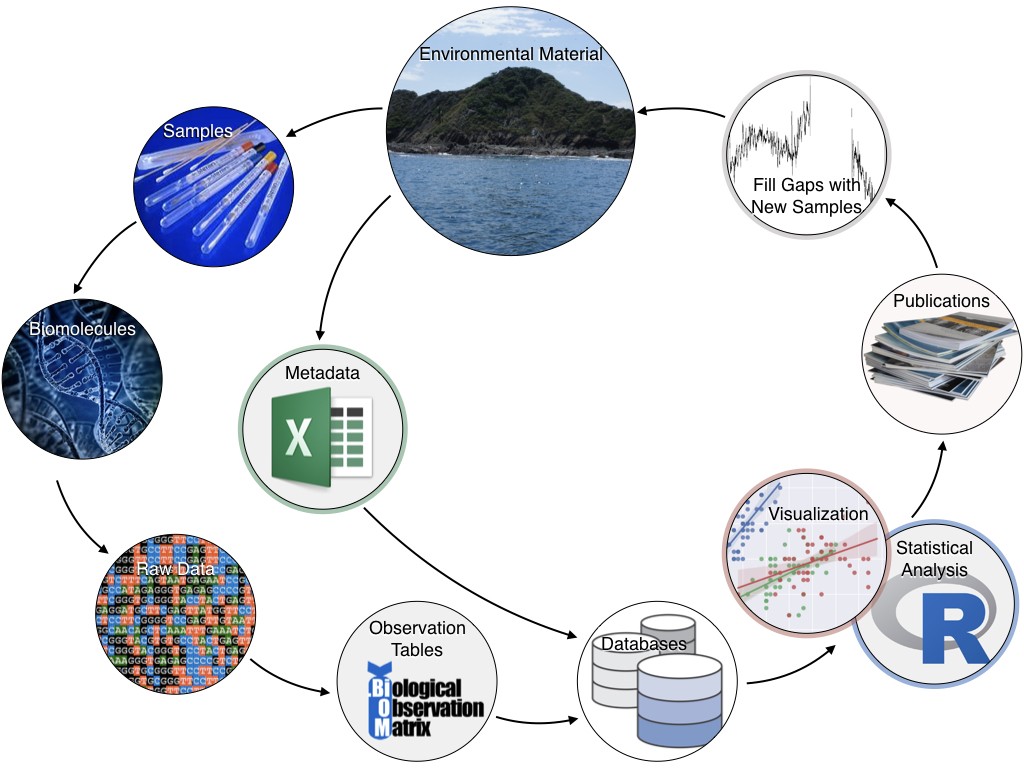
Interpretación de los datos ómicos con la bioinformática
Las técnicas basadas en el genoma mejoran nuestra capacidad de caracterizar y supervisar los ecosistemas, incluida la caracterización de entornos con potencial comercial. Algunos ejemplos son las firmas genómicas que marcan los recursos naturales, como las reservas de petróleo y gas, las enzimas con potencial comercial, como las farmacéuticas o las de biorremediación, los microbiomas que controlan la salud de las especies silvestres y de acuicultura con valor comercial, y los indicadores de estrés antropogénico que pueden pronosticar la productividad ambiental degradada.
Sin embargo, nuestra capacidad para suministrar conocimientos bioinformáticos no ha seguido el ritmo de la generación de datos de secuencias, lo que ha creado un retraso en los datos y ha dificultado la transición de los datos recogidos a información procesable. Para subsanar esta carencia, el AOML ha estado trabajando para desarrollar la capacidad bioinformática, que es importante para el éxito de todos los proyectos "ómicos". El AOML ha asegurado servidores dedicados al análisis bioinformático, ha contratado a jóvenes científicos para que ayuden en el análisis y ha creado grupos de usuarios (locales y de toda la NOAA) para proporcionar apoyo.
Para facilitar el análisis de los conjuntos de datos de amplicones, se creó una línea de producción bioinformática ("Tourmaline") y se añadió a la cuenta GitHub del AOML. Este flujo de trabajo automatizado de amplicones utiliza QIIME 2 y Snakemake y puede ejecutar Deblur (single-end) o DADA2 (single-end y paired-end); los pasos se pueden ajustar manualmente si es necesario.
Publicaciones y referencias
Thompson, L.R., M.F. Haroon, A.A. Shibl, M.J. Cahill, D.K. Ngugi, G.J. Williams, J.T. Morton, R. Knight, K.D. Goodwin y U. Stingl. (2019). SAR11 del Mar Rojo y Prochlorococcus single-.
Los genomas de las células reflejan pangenomas distribuidos globalmente. Appl Environ Microbiol, https://doi.org/10.1128/AEM. 00369-19. Franzosa, E.A., L.J. McIver, G. Rahnavard, L.R. Thompson, M. Schirmer, G. Weingart, K. Schwarzberg Lipson, R. Knight, J.G. Caporaso, N. Segata y C. Huttenhower. (2018). Perfiles funcionales a nivel de especie de metagenomas y metatranscriptomas. Nat Meth, https://doi.org/10.1038/s41592-018-0176-y. Thompson, L.R., J.G. Sanders, D. McDonald, A. Amir, J. Ladau, K.J. Locey, R.J. Prill, A. Tripathi, S.M. Gibbons, G.Ackermann, J.A. Navas-Molina, S. Janssen, E. Kopylova, Y. Va ́zquez-Baeza, A. Gonza ́lez, J.T. Morton, S. Mirarab, Z.Z. Xu, L. Jiang, M.F. Haroon, J. Kanbar, Q. Zhu, S. Song, T. Kosciolek, N.A. Bokulich, J. Lefler, C.J. Brislawn, G.C. Humphrey, S.M. Owens, J. Hampton-Marcell, D. Berg-Lyons, V. McKenzie, N. Fierer, J.A. Fuhrman, A. Clauset, R.L. Stevens, A. Shade, K.S. Pollard, K.D. Goodwin, J.K. Jansson, J.A. Gilbert, R. Knight & The Earth Microbiome Project Consortium. (2017). Un catálogo comunitario revela la diversidad microbiana multiescala de la Tierra. Nature 551:457-463,https://doi.org/10.1038/ naturaleza24621. Goodwin, K.D., L.R. Thompson, B. Duarte, T. Kahlke, J.C. Marques & I. Cac ̧ador. (2017). DNA sequencing as atool to monitor marine ecological status. Front Mar Sci 4:107, https://doi.org/10.3389/fmars.2017.00107. Amir, A., D. McDonald, J.A. Navas-Molina, E. Kopylova, J. Morton, Z.Z. Xu, E.P. Kightley, L.R. Thompson, E.R. Hyde, A. González y R. Knight. (2017). Deblur resuelve rápidamente los patrones de secuencia de la comunidad de un solo nucleótido. mSystems 2(2):e00191–16, https://doi.org/10. 1128/mSystems.00191-16.
¿Buscando literatura? Busque en nuestra base de datos de publicaciones.
















